KDEを使った相互情報量の推定
カーネル密度推定(Kernel density estimation)はノンパラメトリックな確率密度関数の推定法である。 相互情報量(Mutual Information)の推定には、これを用いるのが最も基本的な方法である。 確率変数間の相互情報量は次式で定義される。
ご覧のように、積分が出てきているので、まずはこれを潰さなければならない。 ここには前回、前々回で使ったサンプル平均の考え方を用いる。 さらに各確率密度関数を推定する必要があるが、今回は上述のようにカーネル密度推定により達成する。 最終的に、以下のように相互情報量を推定する。
実行結果を以下に示す。これは真の値は出せていないので、簡単な例で解釈する。 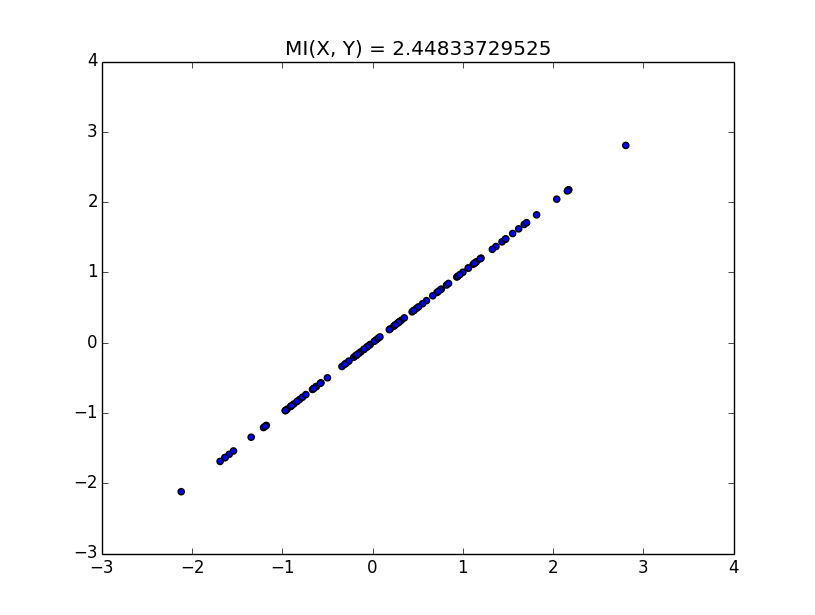
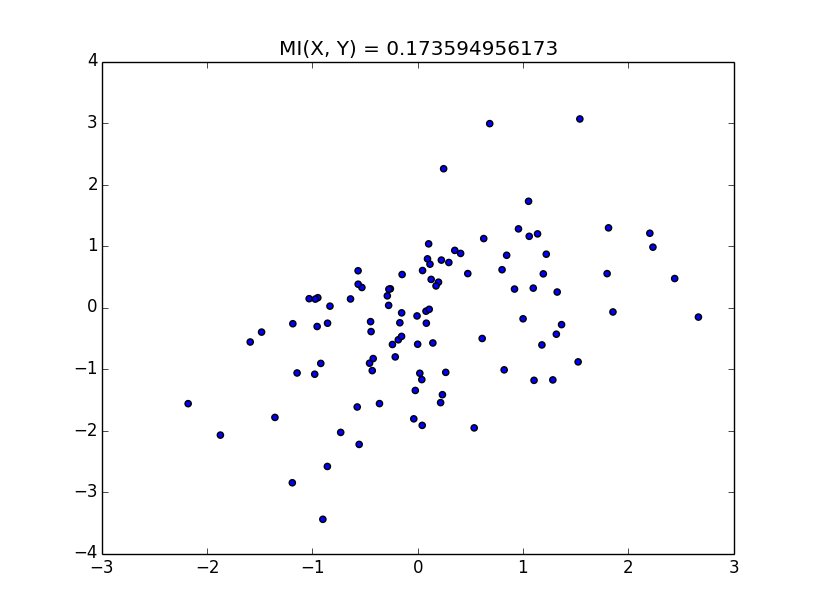
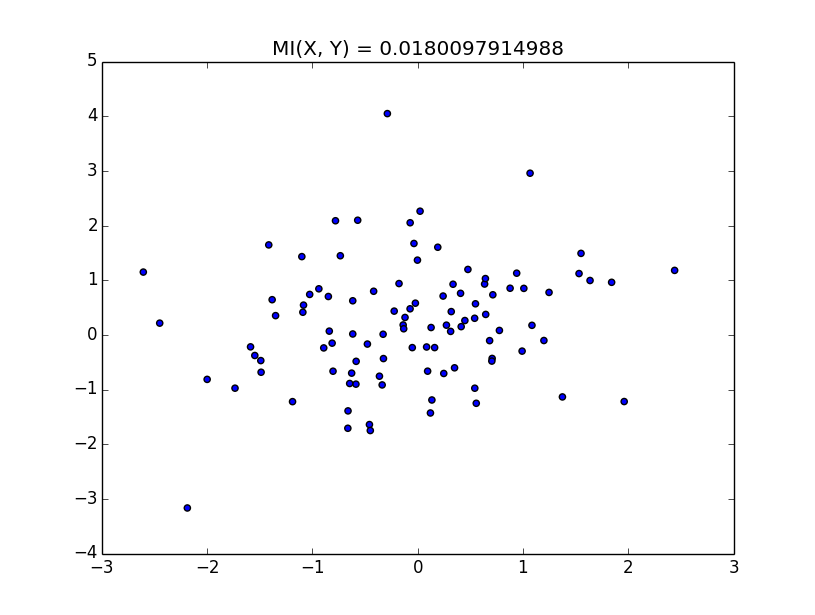 このように、xとyになんらかの相関がある場合、相互情報量は高くなる。 逆になんの相関も無いならば、相互情報量は低くなる。 しかし、単に相関を測るなら相関係数で良いじゃないか、となる。 一般に普通の相関係数は線形な相関しか測れない。 非線形な相関を測りたいならば、相互情報量が有用だ、という話を聞いたことがあるのでその辺りを次回確認したい。 さらに、相関係数の非線形バージョンもあるみたいなので、その辺りも調べて行きたい。 実験に使ったコードを以下に示す。
このように、xとyになんらかの相関がある場合、相互情報量は高くなる。 逆になんの相関も無いならば、相互情報量は低くなる。 しかし、単に相関を測るなら相関係数で良いじゃないか、となる。 一般に普通の相関係数は線形な相関しか測れない。 非線形な相関を測りたいならば、相互情報量が有用だ、という話を聞いたことがあるのでその辺りを次回確認したい。 さらに、相関係数の非線形バージョンもあるみたいなので、その辺りも調べて行きたい。 実験に使ったコードを以下に示す。
import numpy as np
import pylab as pl
from scipy import stats
from sklearn.neighbors import KernelDensity
from sklearn.grid_search import GridSearchCV
from sklearn import metrics
n = 100
mean = [0, 0]
cov = [[1, 0.4], [0.4, 1]]
x = stats.multivariate_normal.rvs(mean=mean, cov=cov, size=n)
params = {"bandwidth": np.logspace(-1, 1, 100)}
kde_xy = GridSearchCV(KernelDensity(), params).fit(x).best_estimator_
kde_x = GridSearchCV(KernelDensity(), params).fit(np.c_[x[:,0]]).best_estimator_
kde_y = GridSearchCV(KernelDensity(), params).fit(np.c_[x[:,1]]).best_estimator_
MI = np.sum(kde_xy.score_samples(x) - kde_x.score_samples(np.c_[x[:,0]]) - kde_y.score_samples(np.c_[x[:,1]]))/n
pl.scatter(x[:,0], x[:,1])
pl.title("MI(X, Y) = {0}".format(MI))
pl.show()